Cybergenetics
Objektive Analyse von DNA-Mischspuren mit Matlab
Fortsetzung des Artikels von Teil 1
Analyse komplexer DNA-Proben mit Matlab
Die TrueAllele-Technologie nutzt Matlab, Signal-Processing-Toolbox und Statistics-Toolbox, um DNA-Mischspuren den einzelnen Verursachern mathematisch zuzuweisen und die jeweiligen Genotypen abzuleiten. Da die Lösung Unsicherheiten birgt, werden den abgeleiteten Genotypwerten Wahrscheinlichkeiten zugewiesen.
Ein DNA-Sequenzer generiert Fluoreszenz-Daten, die mit Hilfe eines Lasers sichtbar gemacht werden. Die Daten werden als eindimensionales Signal dargestellt, das einem mehrfarbigen Multiplex mehrerer Loci entspricht. Das mit Signal-Processing-Toolbox entwickelte TrueAllele-Analysemodell verarbeitet die Signaldaten, um Artefakte zu entfernen. Anschließend werden die Peaks klassifiziert, die Größe und Höhe der Peaks bestimmt und weitere Qualitätsprüfungen durchgeführt.
Nach dieser ersten Analyse interpretiert TrueAllele die Daten mit Hilfe eines Wahrscheinlichkeitsmodells. Dieses Modell umfasst mehrere hundert Variablen, einschließlich der unbekannten Genotypen der Verursacher der Probe, DNA-Quantität, signalverzerrende Amplifikationsartefakte sowie die Unsicherheiten dieser Variablen. Viele dieser Variablen sind hierarchisch, d.h. sie enthalten Untermodelle, die wiederum eigene Parameter und Unsicherheiten aufweisen. Ausgehend von den DNA-Informationen löst TrueAllele das Modell mit Hilfe des statistischen Markow-Chain-Monte-Carlo-Stichprobenverfahrens (MCMC-Verfahren), wobei ein mit Statistics-Toolbox entwickelter Metropolis-Hastings-Algorithmus angewendet wird. Dieser vom Metropolis-Algorithmus abgeleitete Algorithmus ermöglicht es, Folgen von Zufallsvariablen, genauer Markow-Ketten, zu simulieren, die eine gewünschte Verteilung als stationäre Verteilung besitzen, insbesondere in vielen Fällen, wo für die Verteilung Zufallsvariablen nicht direkt simuliert werden können.
Für die Auswertung der DNA-Spur schlägt TrueAllele 100.000 unterschiedliche Kombinationen aus möglichen Werten für Lösungsraumvariablen vor und beurteilt, wie gut jede vorgeschlagene Lösung die DNA-Informationen beschreibt. Anschließend berechnet die auf Matlab basierende Software die Wahrscheinlichkeitsdichten, um eine Wahrscheinlichkeitsverteilung für die möglichen Lösungen vorzunehmen. Lösungen, die die untersuchten Daten am genauesten beschreiben, erhalten eine höhere Wahrscheinlichkeit, während weniger guten Beschreibungen eine geringere Wahrscheinlichkeit zugewiesen wird.
Bei manchen Proben ist es möglich, die Mischspur mathematisch in Komponenten nach ihrer jeweiligen Quelle zu trennen, wobei jedem Genotyp eine hohe Wahrscheinlichkeit zugewiesen wird. Bei anderen Proben sind die Ergebnisse weniger sicher. Die Wahrscheinlichkeiten der sich ergebenden Genotypen sind in diesem Fall diffuser. Unabhängig davon stellt die Genotypantwort jedoch eine Wahrscheinlichkeitsverteilung dar, die objektiv und ausschließlich aus dem Beweismaterial abgeleitet wurde.
Bei Übereinstimmung der DNA-Spur und dem Genotyp eines Verdächtigen können Analytiker mit Hilfe des True-Allele-Modells eine DNA-Übereinstimmungsstatistik berechnen. Um diese Übereinstimmungsstatistik („Likelihood-Quotient“) zu erstellen, vergleicht das Matlab-Programm einen aus dem Beweismaterial abgeleiteten Genotyp mit einem Referenzgenotyp des Verdächtigen. Um eine Beeinflussung der Auswertung auszuschließen, wird dieser Vergleich erst durchgeführt, nachdem die computergestützte Ableitung des Genotyps abgeschlossen wurde.
Die Übereinstimmungsberechnung umfasst einen dritten Genotyp, der eine zufällige Bevölkerungsgruppe darstellt und den Nenner bereitstellt, der notwendig ist, um die Wahrscheinlichkeit einer Übereinstimmung mit einer zufälligen Übereinstimmung zu vergleichen. Das mathematische Ergebnis wird in Klartext formuliert und ermöglicht Forensikern, eine klare Aussage zu treffen, z.B. „die Wahrscheinlichkeit einer Übereinstimmung des Beweismaterials mit dem Verdächtigen ist eine Milliarde mal höher als eine zufällige Übereinstimmung“.
Benutzeroberfläche und Datenbankunterstützung
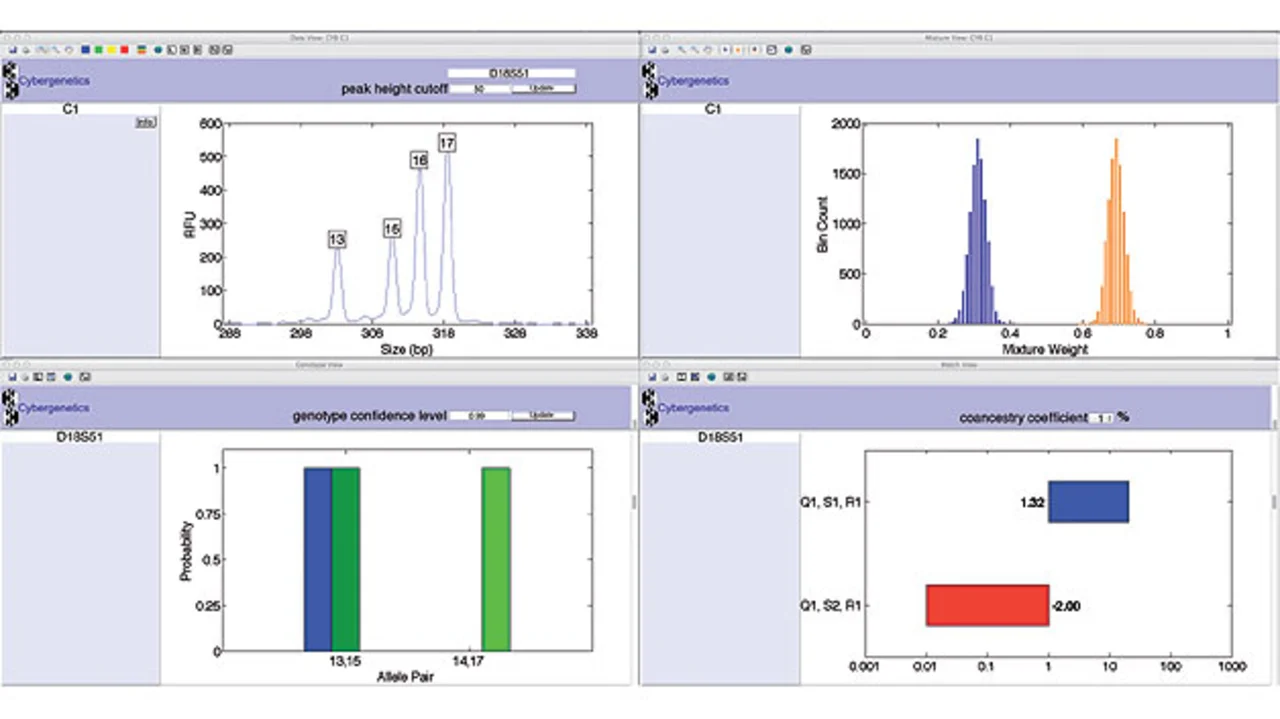
Um die mit TrueAllele gewonnenen Ergebnisse in für Experten, Anwälte und Richter aussagekräftigen Bildern darzustellen, hat Cybergenetics mit Hilfe von Matlab ein sogenanntes Visual User Interface Tool (VUIer) entwickelt. Das VUIer stellt die Schlüsselvariablen bildhaft dar, darunter DNA-Informationen, Gewichtung der Mischspuren, Genotyp-Wahrscheinlichkeiten des Verursachers und Übereinstimmungsgrad (Bild 3). VUIer ermöglicht eine für Lehrzwecke nützliche „Was-wäre-wenn-Analyse“ alternativer Genotypen und Mischmöglichkeiten. Die Benutzeroberfläche berechnet Likelihood-Quotienten und Konfidenzintervalle und ermöglicht die Erstellung eines Berichts zur DNA-Übereinstimmung.
Cybergenetics nutzte den Matlab-Compiler, um eine Standalone-Applikation des VUIer Client zu erzeugen. Der VUIer Client ist plattformübergreifend und kann sowohl unter Mac OS X als auch unter Microsoft Windows ausgeführt werden. Der TrueAllele-Server kann MCMC-Genotypberechnungen parallel auf mehreren Computern unter Linux ausführen. Matlab ermöglicht die Entwicklung in einer einzigen Umgebung, die auf drei verschiedenen Plattformen bereitgestellt werden kann.
Der Zugriff der VUIer Client Software auf den TrueAllele-Datenbankserver erfolgt über Database-Toolbox. Diese PostgreSQL-Datenbank dient als Repository für DNA-Daten, Auswertungsanfragen und Ergebnisse. Die zentrale Serverdatenbank koordiniert den Betrieb des Systems eigenständig mit Hilfe eines eigenen, mit Matlab programmierten Supervisory-Expertensystems.
Der Autor:
| Dr. Mark Perlin |
|---|
| ist Gründer, Chef-Wissenschaftler und CEO von Cybergenetics. Seit 20 Jahren entwickelt er Computer-Modelle für die Interpretation von DNA-Informationen. |
info@cybgen.com
- Objektive Analyse von DNA-Mischspuren mit Matlab
- Analyse komplexer DNA-Proben mit Matlab